Array-Technologien
 Die molekularen Fragestellungen an den
Pathologen beziehen sich zunehmend nicht nur auf das Vorhandensein oder die Abwesenheit bestimmter Gene, sondern auf mehrere Gene gleichzeitig oder den gleichzeitigen Nachweis verschiedener Genvarianten. Hier ist die Array-Technologie eine
vielseitige Methode, die zunehmend in die Diagnostik Einzug hält. Sie stellt dabei eine Hochdurchsatz-Technologie dar, mit der der Nachweis von Genen auf DNA-Ebene möglich ist, aber auch mittelbar die Stärke der Genexpression
auf RNA-Ebene gemessen werden kann.
Die molekularen Fragestellungen an den
Pathologen beziehen sich zunehmend nicht nur auf das Vorhandensein oder die Abwesenheit bestimmter Gene, sondern auf mehrere Gene gleichzeitig oder den gleichzeitigen Nachweis verschiedener Genvarianten. Hier ist die Array-Technologie eine
vielseitige Methode, die zunehmend in die Diagnostik Einzug hält. Sie stellt dabei eine Hochdurchsatz-Technologie dar, mit der der Nachweis von Genen auf DNA-Ebene möglich ist, aber auch mittelbar die Stärke der Genexpression
auf RNA-Ebene gemessen werden kann.
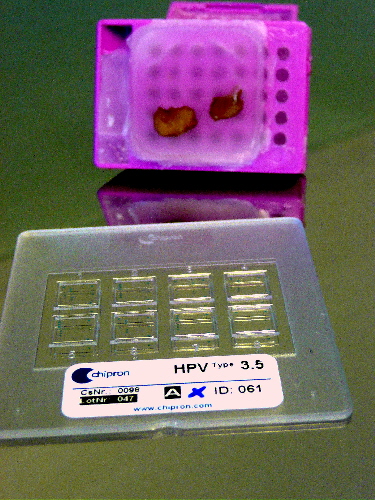
Die Technik basiert auf der Immobilisierung von zahlreichen verschiedenen Oligonukleotid-Sonden als winzige Spots auf einem Träger (zumeist ein speziell behandelter Objektträger, wie er in der Histologie verwendet wird). Die Spots werden als zweidimensionales Gitter aufgebracht und können durch Miniaturisierung eine extrem hohe Dichte von über 10000 Gensonden pro Objektträger erreichen. Für klinische Fragestellungen werden aber meist Arrays mit unter 100 Sonden verwendet (sog. Low density DNA-Chips).
Die Proben-DNA wird zunächst markiert und dann auf den Array hybridisiert. Die Markierung erfolgt mit Fluorezenzfarbstoffen oder immunologisch nachweisbaren Haptenen (Biotin, Digoxigenin) durch eine vorgeschaltete Reaktion (z.B. eine PCR).
DNA-Arrays werden aktuell eingesetzt zur Erreger-Bestimmung (z.B. HPV-Typisierung, Mykobakterien, Pilze), zur Mutationsanalyse (z.B. k-ras), zur Alleltypisierung (CYP2D6) und zur Genexpressionsanalyse (Oncotype DX, Mammaprint).


© 2001-2014
Dr. A. Turzynski
Gemeinschaftspraxis
Pathologie
Lübeck